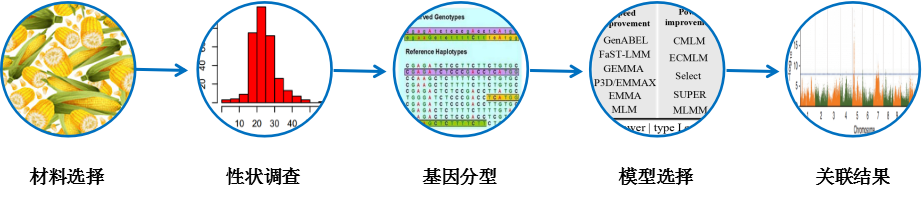
全基因组关联分析(GWAS)
GWAS介绍
全基因组关联分析是对多个个体在全基因组范围的遗传变异多态性进行检测,获得基因型,进而将基因型与可观测的性状,即表型,进行群体水平的统计学分析,根据统计量或P值筛选出有可能影响该性状的遗传变异。
材料选择
不同品种、亚种、地方种/种质库/混合家系/野生资源

不同品种、亚种、地方种

半同胞家系/全同胞家系/野生资源

分析流程
重测序-GWAS
通过全基因组大样本重测序对动植物重要种质资源进行全基因组的基因型鉴定,并与关注的表型数据进行全基因组关联分析(GWAS),进而找到与关注表型相关的SNP位点,定位与性状相关基因,为后续动植物的育种提供科学理论依据。
分析内容
- 1、测序数据质控
- 2、与参考 基因组比对
- 3、全基因组变异检测
- 4、遗传进化分析
- 5、连锁不平衡分析
- 6、全基因组关联分析: a)SNP与性状关联分析 b)候选基因功能注释
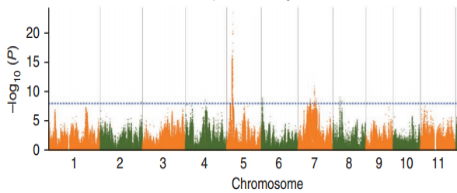
全基因组关联分析
SLAF-GWAS
SLAF-GWAS是应用简化测序的方式,以基因组中检测到的单核苷酸多态性(single?nucleotide?ploymorphism,SNP)为分子遗传标记,进行全基因组水平上的对照分析或相关性分析,统计分析每个变异与目标性状之间的关联性大小,通过比较发现影响复杂性状的基因变异的一种策略。
分析内容
- 测序数据质控
- SNP检测与注释
- 遗传进化分析
- 全基因组关联分析:1)SNP与形状关联分析2)候选基因功能注释
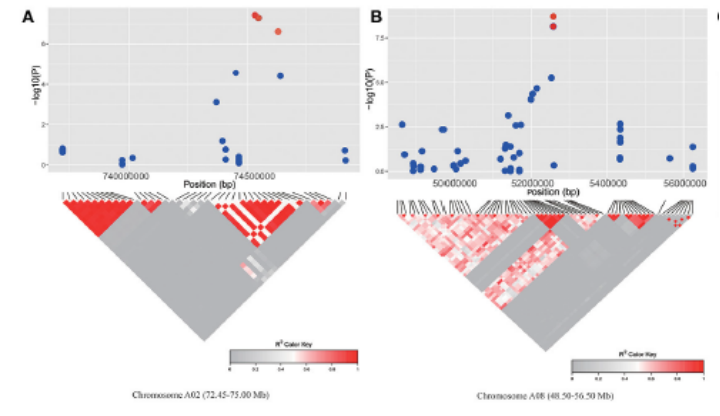
LD-Block分析
项目经验丰富,硕果累累!
百迈客自2009年成立以来,经过多年的厚积薄发,在群体GWAS研究方面已完成上百个物种的项目,不完全统计,协助研究者发表文章70余篇,累计影响因子达340+,其中包含多篇Nature Communications、Molecular?Plant Plant?Biotechnology?Journal及The?Plant?Journal等国际等级期刊。
丰富的项目案例
优秀的分析人员
火箭般的进展速度
数据深度挖掘
案例文章
- Nature Communications百迈客合作文章油菜重测序-GWAS详解2019年03月11日,Nature Communications在线发表了西南大学农学与生物科技学院与北京百 […]阅读更多
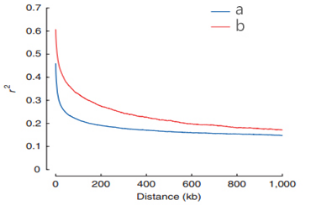
连锁不平衡衰减(LD?decay)分析
在某一群体中,不同座位上某两个基因同时遗传的频率明显高于预期的随机频率的现象,称连锁不平衡?(linkage?disequilibrium)?。自然群体中连锁强度以D’或r2表示,D’或r2越接近于1,代表连锁关系越强。一般用r2大于0.1的数据作为连锁不平衡的衰减(LD?decay)的数值,LD-decay越长,代表物种的SNP间发生重组的概率越小,LD-decay越短,代表物种的SNP间发生重组的概率越大。
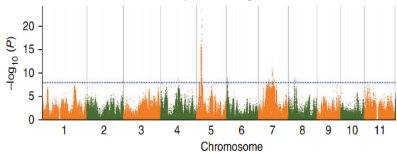
全基因组关联分析
基于SNP,利用TASSEL软件的混合线性模型(compressed?MLM)得到关联值,公式计算为:y=?Xα+Qβ+Kμ+e。其中,通过admixture软件计算样品群体结构Q,通过SPAGeDi软件计算样品间亲缘关系K,X为基因型,y为表型,最终每个SNP位点都能得到一个关联值。最终将性状关联到基因组XX号染色体上。
LD-Block分析
基于SNP,利用Haploview单体型预测软件,预测得到单体型块(趋向于整体遗传的基因序列,Block)。Block大小分布体现了在整个基因组范围内趋向于整体往下遗传的基因序列的长度,block越小,说明基因组重组越多。
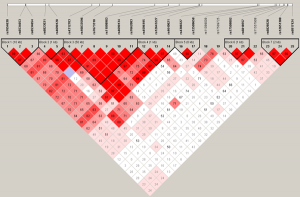

LD-Block的实际应用
一般情况下,全基因组关联分析的曼哈顿图常常与染色体位置、LD-Block图进行联合分析,根据显著关联的SNP位点确定注释的染色体区域,同时在该区域进行LD-Block分析,找到与该位点连锁较强的其他SNP位点,并对其所在的基因进行功能注释。
GWAS分析选材原则是什么?
1、保证选取的样本具有足够的代表性;
2、样本中不能有明显的亚群分化(例如生殖隔离等),因为明显分化的群体会使得遗传背景的噪音较大;
3、建议选择几个比较重要且遗传力较高的表型性状作为研究的重点;
4、质量性状尽量为0、1二值性状,并且两类性状的样本数应当尽量相近;
5、数量性状尽量√准量化记录(如抗病性可以量化为发病率、死亡率、存活率、病斑数、病斑面积等,而不是简单的多级衡量),并使表型总体呈近似正态分布;
6、栽培植物可以进行多年多点多重复记录,多年多点的观测结果可以分别进行关联分析,多重复可以取平均值进行关联分析;
7、表型变异丰富、性状有明显的主效位点控制时样本量可以适当减小,推荐200个以上;表型差异较小,多基因控制时样本量应当增大,推荐500个以上。
自然群体GWAS的研究对象有哪些?
非严格遗传群体:
1、种质资源
2、半同胞家系,混合家系
3、MAGIC/NAM家系
4、多个F2/RIL/全同胞家系
5、高杂合类物种:F1群体







 京公网安备 11011302003368号
京公网安备 11011302003368号