微生物多样性测序分析
产品介绍
微生物多样性测序
微生物多样性测序是对环境样品中细菌(16S rDNA)、真菌(18S/ITS)、功能基因等基因的DNA进行特定长度的PCR扩增并对扩增产物进行测序的分析,可实现对环境样品中的优势物种、稀有物种和一些未知物种进行检测,精准解析微生物在种属水平上的组成和丰度情况,是当前研究环境微生物多样性及群落组成差异的重要技术手段。
细菌微生物多样性
16S?rDNA测序:16S?rDNA具有10个保守区和9个高变区。保守区反映生物物种间的亲缘关系,高变区反映物种间的差异。对16S?rDNA某个高变区进行测序,用于研究环境微生物中的群落结构多样性。
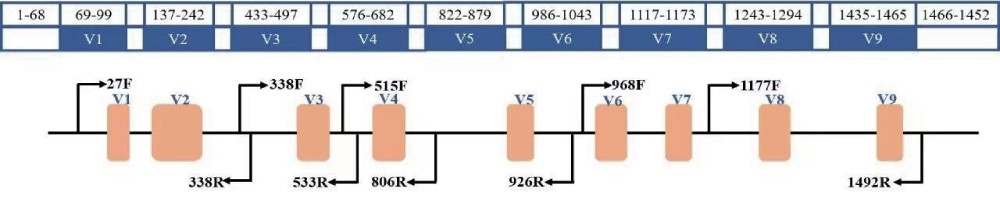
真菌微生物多样性
18S?rDNA测序:18S?rDNA也包含可变区和保守区。对18S?rDNA某个高变区进行测序,用于研究环境样本中真核微生物群落结构多样性。
ITS1测序:ITS分为两个区域:ITS1和ITS2。ITS区域进化速率是18S rDNA的10倍之多。对ITS1和ITS2进行测序,用于研究环境样本中真核微生物群落结构多样性。

功能基因多样性
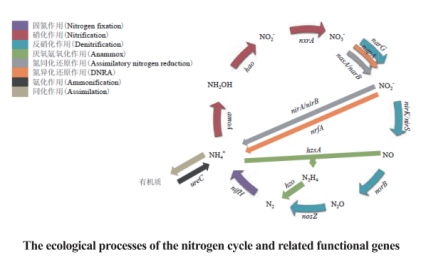
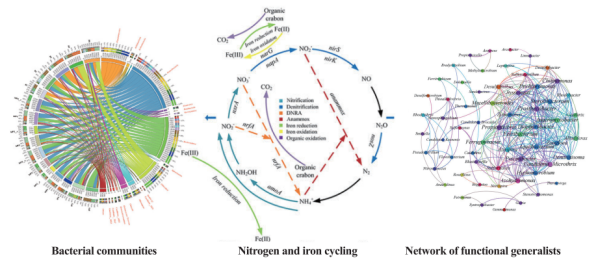
功能微生物是在自然界中由于其功能的重要性而受到广泛关注的一类微生物,如硝化细菌、反硝化细菌、氨氧化细菌、硫酸盐还原菌、固氮菌等,使这些功能细菌发挥这种特定功能的基因就称为功能基因,如AOA、AOB、nifH等。
功能基因测序,是根据复杂环境中具有特殊功能的微生物基因序列设计引物,用该引物进行PCR扩增,建库后进行高通量测序。功能基因测序可有效研究特定环境中的功能微生物物种信息,包括:功能微生物功能差异、分类和丰度等,并与每个采样点的环境因子相结合,可以同时分析环境因子与功能细菌群落结构的关系,因此对复杂环境中的微生物构成研究有重要的指导作用。
技术路线
技术策略与送样要求
测序策略
Illumina NovaSeq测序
?DNA送样要求
DNA浓度≥1ng/ul,总量≥30ng,DNA电泳条带清晰,无降解或者轻度降解。
环境样品取样方法及送样要求
| 环境样品 | 送样要求 | 保存及运输 |
| 普通土壤 | 除去土壤表层未分解的凋落物层、动植物残体、石砾等杂质,将大块的样品捣碎,过2mm筛后,分装至2mL或更大体积的EP管或冻存管中;每管土壤含量大概0.25~0.5g,需保证送样量在1~2g。 | 样本-80℃或液氮中长期保存,干冰运输 |
| 根际土壤 | 收集植物植株,去除根部大块土壤;摇动植株去除附着不紧密的土壤,使用无菌刷子收集根部附着紧密的土壤;随机多点取样5-10g,过2mm筛后,分装至2mL或更大体积的EP管或冻存管中;每管土壤含量大概0.25~0.5g,需保证送样量在1~2g。 | |
| 粪便 | 无菌牙签或粪便取样器截取样品中段内部(避免表层中的肠道膜脱落细胞),外部容易污染且细菌DNA由于接触空气可能有降解。将已取的粪便样品分装至2mL?EP管(无菌)或冻存管(无菌)中,每管粪便量为0.5~2g,每个样品分装2~3管备份。 | |
| 肠道内容物 | 在实验对象死亡后,无菌条件下,取出整个肠道,用无菌解剖刀切取所需肠段的,用无菌手术刀挖取内容物分装至2mL?EP管(无菌)或冻存管(无菌)中,每管组织量为0.5~2g,每个样品分装2~3管备份 | |
| 水体 | 过滤大量低微生物含量的清亮水样用0.22μm?的聚苯醚砜滤膜,每个样本至少1L水样。浑浊水样使用0.22μm滤膜过滤缓慢容易堵塞时,建议使用0.45μm的混合纤维素酯滤膜,每个样本0.5L-1L水样;如果水样中不可溶解的颗粒较多,需要使用2-5μm孔径的滤膜将不可溶解的颗粒杂质滤去,再使用0.22μm或0.45μm的滤膜富集菌体,每个样本0.5L-1L水样。 | |
| 空气 | 开动采样仪(浮游细菌采集器),使被测空气滤过凝胶膜(灭菌),空气中的浮游菌被截留在凝胶膜上(过滤时间越长,收集的空气中的灰尘越多,菌数量越多),收集完成后取下滤膜。 |
产品优势与项目经验
高水平分析团队
丰富的项目经验
全面的分析内容
专业的售后服务
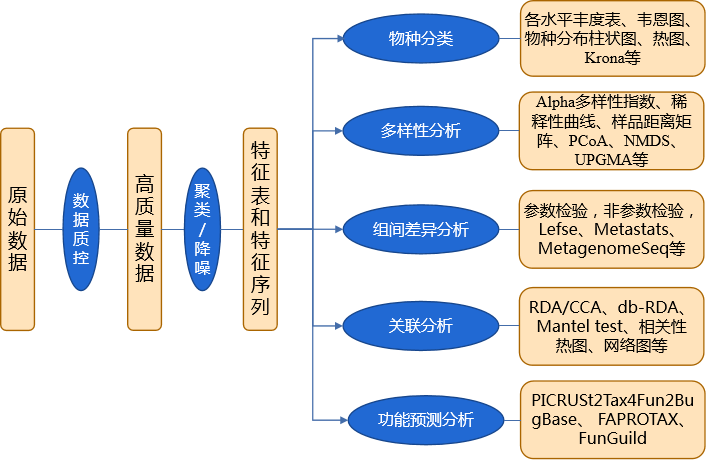
数据质控
数据下机后根据PE reads之间的Overlap关系,将Hiseq测序得到的双端序列数据拼接(Merge)成一条序列Tags,同时对Reads的质量和Merge的效果进行质控,主要包括去除低质量Tags和去嵌合体
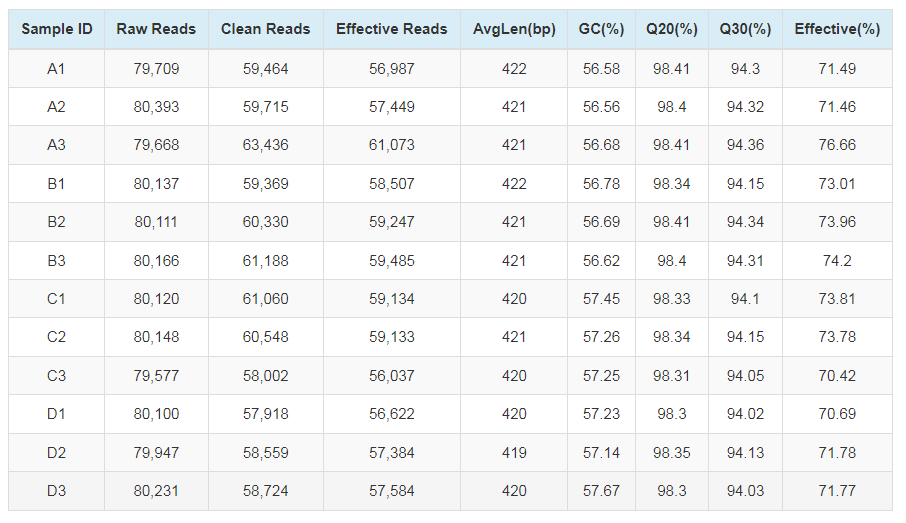
物种注释
根据97%的相似度进行OTU聚类后,基于Silva(细菌)和UNITE(真菌)分类学数据库对OTU进行分类学注释,得到每个OTU对应的物种分类信息,进而在各水平(phylum,class,order,family,genus,species)统计各样品群落组成,绘制样品在各分类学水平下的群落结构柱状图,柱状图展示不同样品中物种的组成和相对丰度。
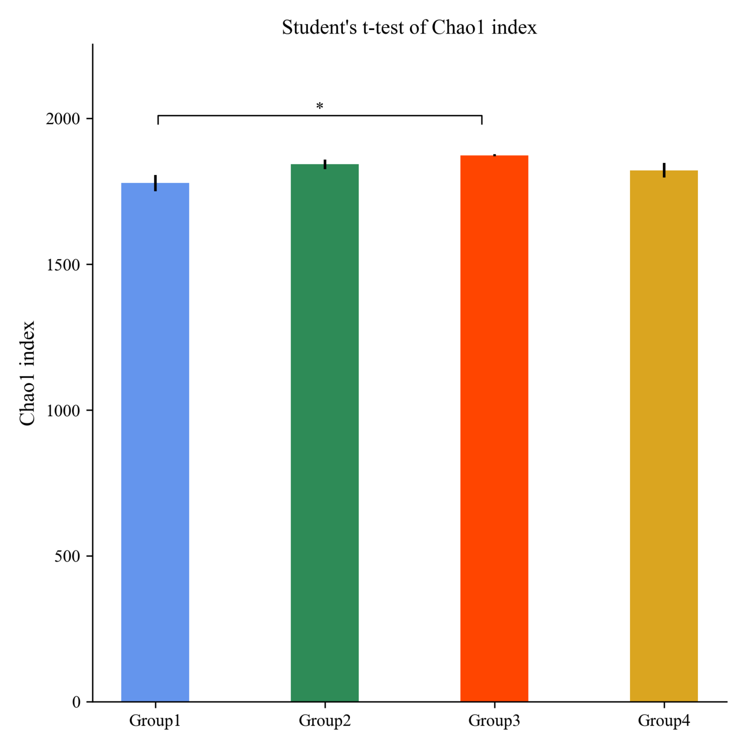
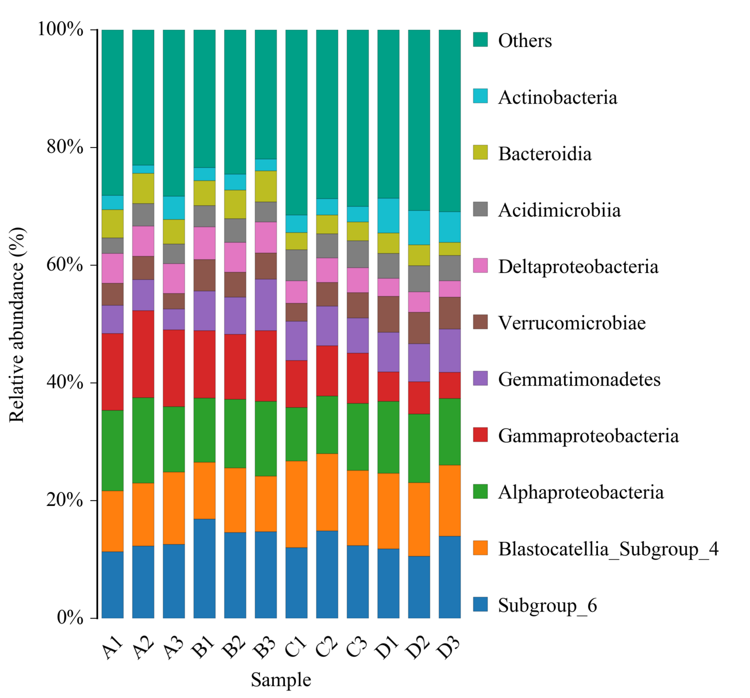
Beta多样性分析
Beta多样性分析主要采用 binary jaccard、 bray curtis、 weighted unifrac(限细菌)、 unweighted unifrac (限细菌)等4种算法计算样品间的距离从而获得样本间的β值。这四个算法主要分为两大类:加权(Bray-Curtis和Weighted Unifrac)与非加权(Jaccard和Unweightde Unifrac)。利用非加权的计算方法,主要比较的是物种的有无;而加权方法,则需要同时考虑物种有无和物种丰度两个问题。样品间距离越小,说明两个群体的物种组成越相似。
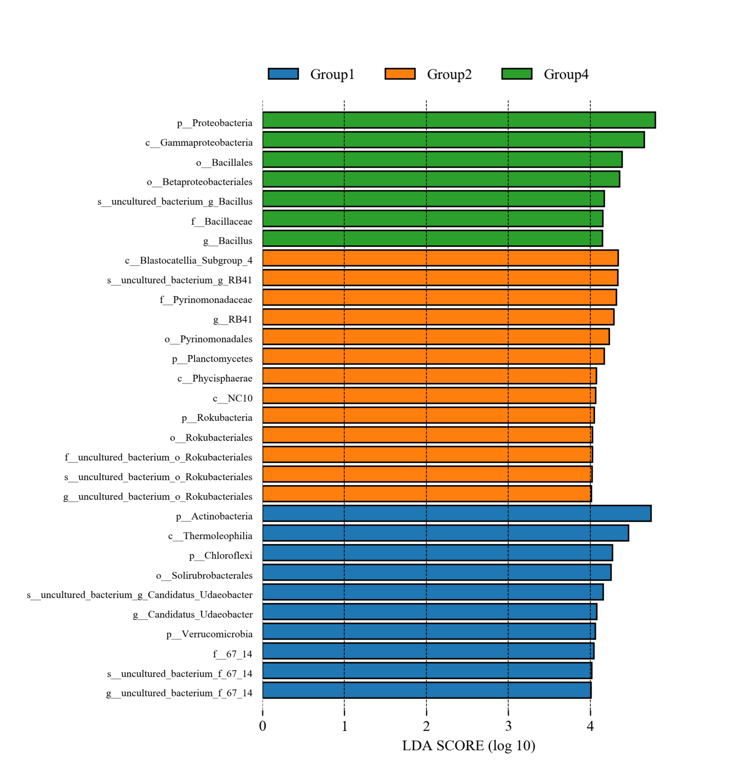
LEfSe分析
LEfSe(Line Discriminant Analysis (LDA) Effect Size)分析主要用于发现不同组间具有统计学差异的Biomarker,根据设定的Biomarker筛选标准(LDA score>4)找出符合条件的Biomarker。
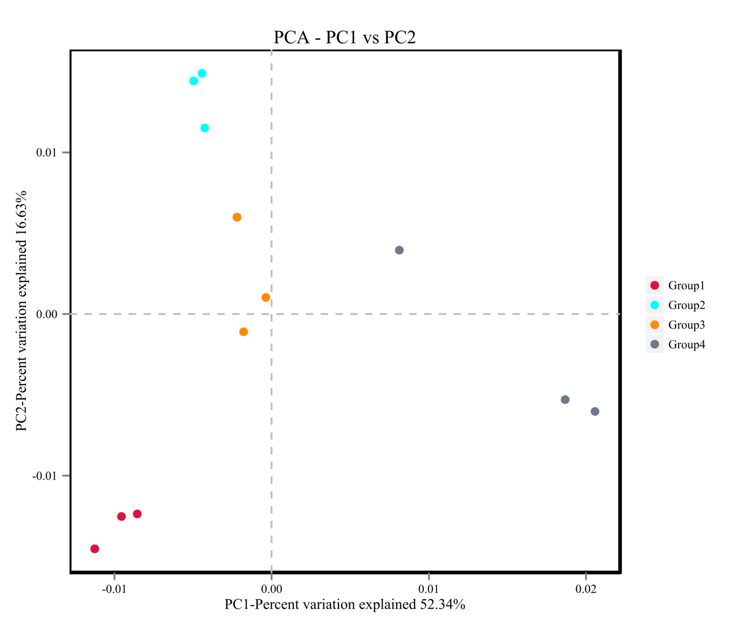
主成分分析(Principal Component Analysis,PCA)
PCA是一种分析和简化数据集的技术,通过将方差进行分解,将多组数据的差异反映在二维坐标图上,通过分析不同样品 OTU(97%相似性)组成可以反映样品的差异和距离,PCA运用方差分解,将多组数据的差异反映在二维坐标图上,坐标轴取能够最大反映方差的两个特征值。图中两个样品距离越近,则表示这两个样品的组成越相似。
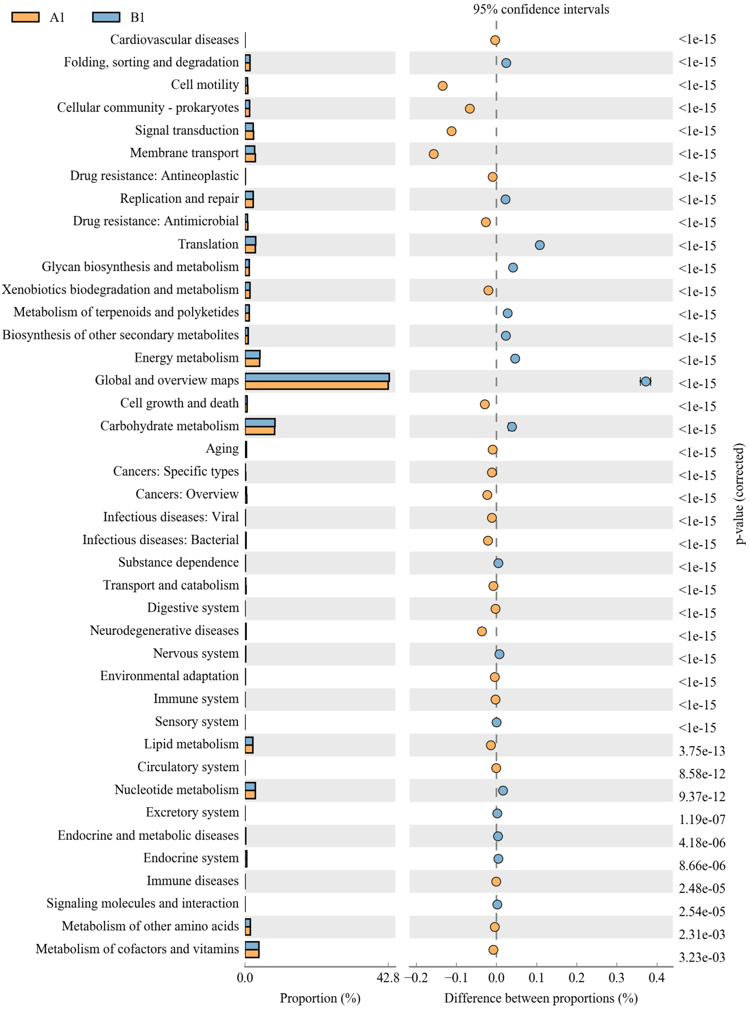
16S功能预测
16S功能预测基于PICRUSt软件通过比对16S测序数据获得的物种组成信息,推测样本中的功能基因组成,从而分析不同样本或分组之间在功能上的差异。

相关性分析
网络图是相关性结果的一种表现形式,根据各个物种在各个样品中的丰度以及变化情况,使用sparcc算法进行相关分析,图中线的粗细代表相关性的强弱;线的颜色表示相关性:红色代表正相关,绿色代表负相关。
成功案例展示
案例一
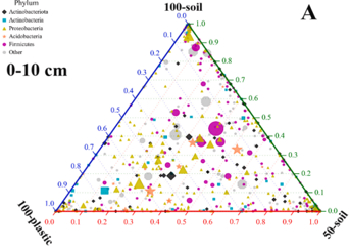
Degradation of polyethylene plastic in soil and effects on microbial community composition
发表时间:2021
合作单位:西北农林科技大学
发表期刊: Journal of Hazardous Materials
研究背景:全球土壤系统中的塑料污染正成为一个严重的全球性问题,对陆地生态系统服务和人类健康构成潜在威胁。为了确定聚乙烯薄膜的降解性和生态效应,本研究以小麦为试材,通过盆栽试验,研究塑料降解与土壤微生物群落组成之间的相互作用。
测序策略: 16S V3+V4 Illumina Hiseq 2500 PE250
研究结论:不同条件下的PE膜具有独特的降解性能,改变了土壤微生物群落组成。此外,在塑料薄膜上形成了一个巨大的独特的微生物群,这可能会改变土壤的基本性质。PE膜可能是一种独特的微生物定殖基质,可能促进其自身降解,改变生物地球化学过程和土壤生态功能。
参考文献: Daofen Huang, Yibo Xu, Fadan Lei, Xiaoqin Yu, Zhuozhi Ouyang, Yanhua Chen, Hanzhong Jia, Xuetao Guo,Degradation of polyethylene plastic in soil and effects on microbial community composition,Journal of Hazardous Materials,2021

案例二
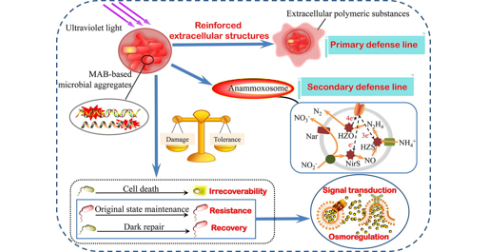
Nitrogen removal mechanism of marine anammox bacteria treating nitrogen-laden saline wastewater in response to ultraviolet (UV) irradiation: High UV tolerance and microbial community shift
发表时间:2021
合作单位:青岛大学
发表期刊: Bioresource Technology
研究背景:海洋厌氧氨氧化菌(MAB)可以自然克服盐胁迫,但其生长速率低、对操作条件敏感仍是应用厌氧氨氧化的挑战。为增强MAB的富集,深入了解紫外线辐照与MAB的关系,将紫外线引入MAB脱氮工艺中,研究MAB在不同紫外线剂量下处理含氮含盐废水的脱氮机理,分析不同紫外线剂量引起的胞外多聚物(EPS)含量、关键酶活性和微生物群落组成及紫外线照射下微生物预测功能的动态探讨。
测序策略: 16S V3+V4 Illumina Hiseq 2500 PE250
研究结论:在处理含氮盐渍废水中,念珠菌对UV-C辐照的耐受性较高,可达12000 mJ/cm2。在此剂量下,微生物丰富度增加,多样性降低,MAB的相对丰度提高了1.2倍。过量的EPS产量和厌氧氨氧化菌的特殊结构是维持紫外辐射下厌氧氨氧化活性的两条防线。
参考文献: Yuanyuan Gao, Jin Li, Huiyu Dong, Zhimin Qiang,Nitrogen removal mechanism of marine anammox bacteria treating nitrogen-laden saline wastewater in response to ultraviolet (UV) irradiation: High UV tolerance and microbial community shift,Bioresource Technology,2021

Beta 多样性分析主要采用 binary jaccard、 bray curtis、 unweighted Unifrac(限细菌)、weighted Unifrac (限细菌)等4种算法计算样品间的距离,那么这四种算法都有什么差别呢?

非加权的计算方法,主要考虑的是物种的有无,即如果两个群体的物种类型都一致,表示两个群体的样本距离最小;加权方法,则同时考虑物种有无和物种丰度两个问题。比如如样品A由3个物种a和2个物种b组成,样品B由2个物种a和3个物种b组成,则通过非加权方法计算,因为样品A与样品B的物种组成完全一致,都只由物种a和b组成,因此它们之间的样本距离为0。但通过加权方法计算,虽然样品A与样品B的物种组成一致,但物种a和b的数目却不同,因此两个群体的β多样性则并非一致。
基于独立OUT的方法认为OTU之间不存在进化上的联系,每个OTU间的关系平等;基于系统发生树计算的方法,会根据16s的序列信息对OTU进行进化树分类, 因此不同OTU之间的距离实际上有“远近”之分。
主成分分析(Principal Component Analysis,PCA)是一种分析和简化数据集的技术,通过将方差进行分解,将多组数据的差异反映在二维坐标图上;主坐标分析法(Principal coordinates analysis,PCoA)是一种与 PCA 类似的降维排序方法。PCoA与PCA的区别在于PCA是基于原始的物种组成矩阵所做的分析,使用的是欧式距离,仅仅比较的是物种丰度的不同,而PCoA首先根据不同的距离算法计算样品之间的距离,然后对距离矩阵进行处理,使图中点间的距离正好等于原来的差异数据,实现定性数据的定量转换。
- J Hazard Mater | 蜜蜂健康告急!江西农大蜜蜂研究所科研新突破蜜蜂,作为自然界最重要的传粉者之一,正面临前所未有的危机!农药的广泛使用被认为是蜜蜂种群下降的主要原因之一。而 […]阅读更多









 京公网安备 11011302003368号
京公网安备 11011302003368号