Pacbio全长转录组测序
全长转录组研究是理解生物机体功能的一个重要途径。传统二代转录组测序无法直接获得单个RNA分子由5ˊ到3ˊ的全部序列。基于PacBio三代测序平台的转录组研究,无需打断,直接读取反转录的全长cDNA,能够有效的获取高质量的单个RNA分子的全部序列,准确辨别二代测序无法识别的同源异构体(isoform)、同源基因、超家族基因或等位基因表达的转录本。
?分析内容
?结果展示
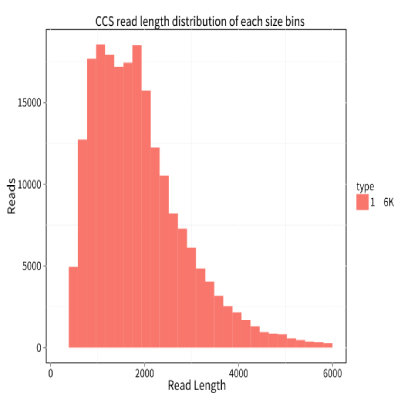
CCS数据展示
PacBio测序仪每个cell含有ZMWs,reads进入ZMW孔中被测序,一个ZMW中含一条的reads(P1)为有效数据。通过有效数据的子序列获得一致序列即为每个单分子测序反应器ZMW的CCS序列。CCS序列是每个单分子测序反应器ZMW里插入序列的最高质量序列。CCS序列可以从一定程度上评估建库质量和SMRT? Cell上样时序列的长度。
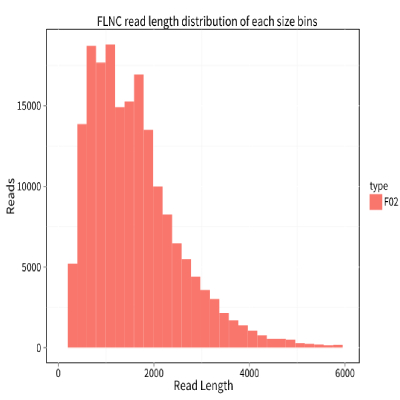
全长转录本数目统计
通过检测CCS序列中是否包含正确的5’引物,3’引物及polyA尾,将序列分成全长序列(包含5’引物,3’引物及polyA尾)和非全长序列。去除CCS序列中cDNA 引物序列及polyA序列获得建库时的插入序列,同时根据建库时两端引物的差别确定链合成方向,将序列分为全长序列和非全长序列、嵌合序列和非嵌合序列。全长序列长度反映了建库时cDNA序列长度,可通过统计全长序列的长度评估建库质量。
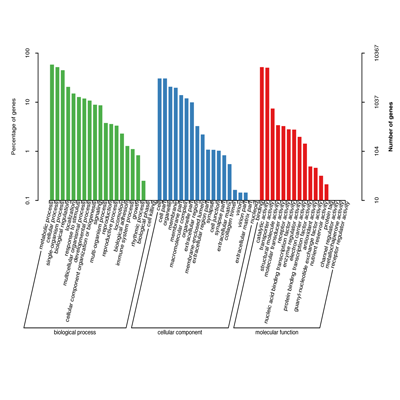
转录本GO注释
GO数据库是GO组织(Gene Ontology Consortium)于2000年构建的一个结构化的标准生物学注释系统,旨在建立基因及其产物知识的标准词汇体系,适用于各个物种。GO注释系统是一个有向无环图,包含三个主要分支,即:生物学过程(Biological Process),分子功能(Molecular Function)和细胞组分(Cellular Component)。
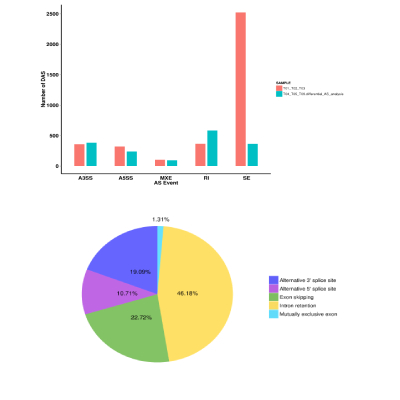
可变剪接分析
基因转录生成的前体mRNA(pre-mRNA),有多种剪接方式,选择不同的外显子,产生不同的成熟mRNA,从而翻译为不同的蛋白质,构成生物性状的多样性。这种转录后的mRNA加工过程称为可变剪接或选择性剪接(Alternative splicing)。可变剪接类型包括:(A) 外显子跳跃;(B) 可变转录终止位点;(C) 可变外显子;(D) 可变转录起始位点;(E) 内含子保留。百迈客使用Astalavista软件获取每个样品存在的可变剪接类型。结合RNA-Seq数据可以使用rMATS进行不同分组间的差异可变剪接分析。
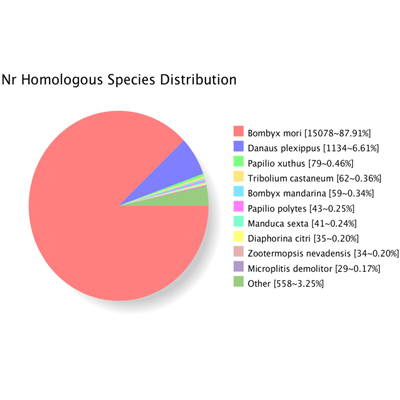
转录本NR注释
Nr数据库是NCBI中的非冗余蛋白质数据库,包含了Swissprot、PIR(Protein Information Resource)、PRF(Protein Research Foundation)、PDB(Protein Data Bank)蛋白质数据库及从GenBank和RefSeq的CDS数据翻译过来的蛋白质数据信息。通过序列比对寻找同源物种,并进行注释。
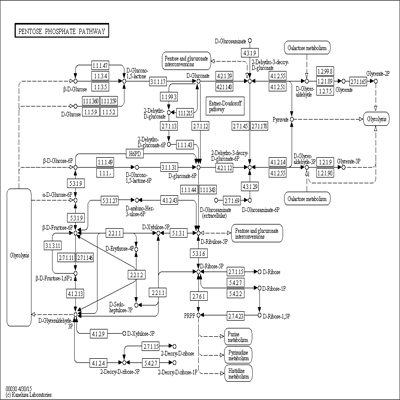
转录本KEGG注释
在生物体内,不同的基因产物相互协调来行使生物学功能,对表达基因的通路(Pathway)注释分析有助于进一步解读基因的功能。KEGG(Kyoto Encyclopedia of Genes and Genomes)是系统分析基因功能、基因组信息数据库,它有助于研究者把基因及表达信息作为一个整体网络进行研究。
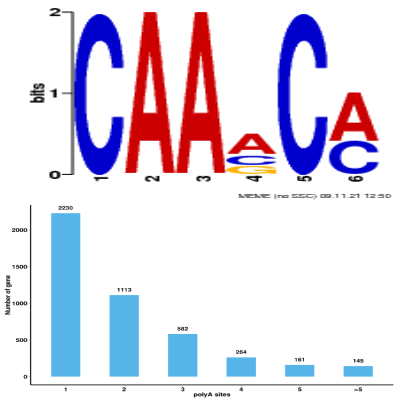
可变多聚腺苷酸化
多聚腺苷酸化是指多聚腺苷酸与信使RNA(mRNA)分子的共价链结。在蛋白质生物合成的过程中,这是产生准备作翻译的成熟mRNA的方式的一部份。在真核生物中,多聚腺苷酸化是一种机制,令mRNA分子于它们的3’端中断。多聚腺苷酸尾(或聚A尾)保护mRNA,免受核酸外切酶攻击,并且对转录终结、将mRNA从细胞核输出及进行翻译都十分重要。在原核生物中,前体mRNA的可变多聚腺苷酸化(alternative polyadenylation,APA)可能贡献于转录组多样性,基因组的编码能力以及基因的调控机制。百迈客采用TAPIS pipeline来对全长非嵌合序列(FLNC)进一步分析以识别APA。
成功案例
- 文献解读|利用Pacbio Iso-Seq测序技术发现水稻非生物胁迫下的新转录本和新基因文章题目:Utilizing PacBio Iso-Seq for Novel Transcript and […]阅读更多
PacBio全长转录组数据处理中各个术语的概念
答:1)Polymerase Read:酶聚合序列,DNA聚合酶以SMRTbell?环状模板链合成的核酸序列,可用于测序过程中每轮(run)的质控。Polymerase reads经过滤后仅剩余高质量片段,包含接头序列和通过环状模板链合成的含多个序列的拷贝。
(2)Subread:每个聚合酶序列(polymerase read)可以分割成一个或多个子序列(Subread),subread是聚合酶以SMRTbell? 一条模板链经过一轮(passes)合成的,不包括接头序列。每个subread包含质量值和相关酶活参数。
(3)number of full passes:指原始序列中存在两端均含有SMRTbell? 接头(adapter,图中黑色区域)的子序列(接头间的序列)个数。
(4)Circular Consensus (CCS) Read:根据需求筛选原始序列中满足最小full pass数为 1,最低序列准确度为 0.9 的原始序列,通过子序列获得一致序列即为每个单分子测序反应器ZMW的CCS序列。CCS序列是每个单分子测序反应器ZMW里插入序列的最高质量序列。CCS序列可以从一定程度上评估建库质量和SMRT? Cell上样时序列的长度。
(5)Full-Length (FL) Read versus Non-Full-Length (nFL) Read:定义两端同时含有3’引物和5’引物,及3’引物前含有polyA尾(可选)的序列称为全长序列(Full-Length(FL) Read)。 5’或3′ primer 可以是Clontech或其他全长cDNA建库引物,或基因特异性的RT-PCR引物。反之,则为非全长序列(non-full-lengthread)。
(6)Full-Length non-chimericRead (FLNC):建库过程中因接头浓度或SMRTbell浓度过低造成两个cDNA模板链直接相连而生成的嵌合序列称为人工嵌合序列,如下图所示。全长序列中的非嵌合序列称为全长非嵌合序列。
PacBio全长转录组是否可以进行定量?
答:PacBio全长转录组由于单cell产出有限,所以无法完成基因定量,需要结合二代转录组(RNA-seq)的数据联合分析,才可以进行基因水平和转录本水平的定量,通常可以选择全长转录组2+3的联合分析产品进行分析。
PacBio全长转录组的优势是什么?
答:
1、无需打断,可直接获得从5’端到3’端的全长转录本序列
2、转录本序列准确性高,连续性、完整性更好
3、准确鉴定转录本水平的结构变异,如可变剪接、融合基因等
4、结合二代转录组测序,同时完成基因水平和转录本水平的准确定量
5、物种适应性高,有无参考基因组均可以进行分析
- Plant J | ATAC-seq助力院士团队在柑橘无融合生殖和FhRWP功能探索中取得新进展ATAC-seq(染色质转座酶可及性测序)是一种高效解析染色质开放区域的技术,通过Tn5转座酶靶向切割松散染色 […]阅读更多
- 【项目文章】Nature Genetics | 国内科学家团队在葡萄属超级泛基因组研究中获得重大突破2025年02月26日,潍坊现代农业山东省实验室、北京大学现代农业研究院叶文秀研究员和郭立研究员团队在国际期刊 […]阅读更多
- Nature Communications | 全长转录组助力重庆医科大学成功解析可变剪切新机制全长转录组是指借助三代测序平台对完整转录本进行不打断的全长测序,序列准确性远远高于需要进行打断测序的二代常规转 […]阅读更多








 京公网安备 11011302003368号
京公网安备 11011302003368号